Metodutveckling
Beskrivning
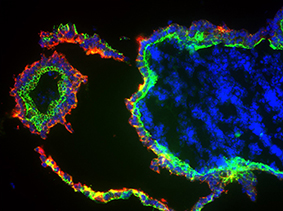
Metoder och teknologier för cellanalys är en hörnsten inom forskning och sjukvård, vilket underlättar forskare att svara på frågor om sjukdomars uppkomst och utveckling, samt hjälper läkare att ställa en korrekt diagnos. Under de senaste två decennierna har utvecklat flera mikroskopibaserade metoder; såsom in situ PLA, ProxHCR och MolBoolean. Dessa metoder gör det möjligt att se protein-interaktioner och post-translationella modifieringar i fixerade celler och vävnader, vilket är avgörande för studier och förståelse av cellsignalering. I våra metoder använder vi DNA som reportermolekyl och metoderna är baserade på naturligt förekommande enzymer. Vår nuvarande forskning utforskar möjligheten att gå bortom gränserna för vad naturen kan ge, att designa enzymer som inte kan skapas genom evolution. Vi har nyligen konstruerat ett DNA-polymeras som ger unika möjligheter för utveckling av nya metoder för medicinsk forskning.
Utvalda publikationer
Publikationer och bibliometri
Google scholar
https://scholar.google.se/citationsuser=XigDIwUAAAAJ&hl=en&oi=ao
Scopus:
https://www.scopus.com/authid/detail.uri?authorId=57204274888
Bivehed E, Hellman B, Wenson L, Stenerlöw B, Söderberg O & Heldin J
”Visualizing DNA single- and double-strand breaks in the Flash comet assay by DNA polymerase assisted end-labelling”
Nucleic Acid Res, 2024, in press
Raykova D, Kermpatsou D, Malmqvist T, Harrison PJ, Rubin Sander M, Stiller C, Heldin J, Leino M, Ricardo S, Klemm A, David L, Spjuth O, Vemuri K, Dimberg A, Sundqvist A, Norlin M, Klaesson A, Kampf C & Söderberg O
“A method for Boolean Analysis of Protein Interactions at a Molecular Level”
Nature Communications, 2022, 13(1):4755
Koos B, Cane G, Grannas K, Löf L, Arngården L, Heldin J, Clausson CM, Klaesson A, Hirvonen MK, Souza de Oliviera FM, Talibov V, Pham N, Auer M, Danielson H, Haybaeck J, Kamali-Moghaddam M & Söderberg O
“Proximity dependent initiation of hybridization chain reaction”
Nature Communications, 2015, 6, 7294
Leuchowius KJ, Clausson CM, Grannas K, Erbilgin Y, Botling J, Zieba A, Landegren U & Söderberg O.
”Parallel visualization of multiple protein complexes in individual cells in tumor tissue”
Mol Cell Proteomics, 2013, 12(6):1563-71.
Weibrecht I, Lundin E, Kiflemariam S, Mignardi M, Grundberg I, Larsson C, Koos B, Nilsson M & Söderberg O.
”In situ detection of individual mRNA molecules and protein complexes or post-translational modifications using padlock probes combined with the in situ proximity ligation assay”
Nature Protocols, 2013, 8(2): 355-72.
Weibrecht I, Gavrilovic M, Lindbom L, Landegren U, Wählby C & Söderberg O.
”Visualising individual sequence-specific protein-DNA interactions in situ”
N Biotechnol, 2012, 29(5): 589-598.
Clausson CM, Allalou A, Weibrecht I, Mahmoudi S, Farnebo M, Landegren U, Wählby C & Söderberg O.
”Increasing the dynamic range of in situ PLA”
Nature Methods, 2011, 8(11): 892-893.
Larsson C, Grundberg I, Söderberg O & Nilsson M.
”In situ detection and genotyping of individual mRNA molecules”
Nature Methods, 2010, 7(5): 395-397.
Jarvius M, Paulsson J, Weibrecht I, Leuchowius KJ, Andersson AC, Wählby C, Gullberg M, Botling J, Sjöblom T, Markova B, Östman A, Landegren U & Söderberg O.
”Detection of PDGF-receptor-β phosphorylation using a generalized proximity ligation method”
Mol Cell Proteomics, 2007, 6(9): 1500-1509.
Söderberg O, Gullberg M, Jarvius M, Ridderstråle K, Leuchowius KJ, Jarvius J, Wester K, Hydbring P, Bahram F, Larsson LG & Landegren U.
”Direct observation in situ of individual endogenous protein complexes by proximity ligation”
Nature Methods, 2006, 3(12): 995-1000.
Doktorsavhandlingar från gruppen
2010
Karl-Johan Leuchowius ” High Content Analysis of Proteins and Protein Interactions by Proximity Ligation”
http://uu.diva-portal.org/smash/get/diva2:301950/FULLTEXT01.pdf
2011
Irene Weibrecht “Visualizing Interacting Biomolecules In Situ”
http://uu.diva-portal.org/smash/get/diva2:410507/FULLTEXT01.pdf
2012
Agata Zieba “Application of Proximity Ligation Assay for Multidirectional Studies on Transforming Growth Factor Receptor-β Pathway”
http://uu.diva-portal.org/smash/get/diva2:513327/FULLTEXT01.pdf
2014
Carl-Magnus Clausson “Making Visible the Proximity Between Proteins”
http://uu.diva-portal.org/smash/get/diva2:695615/FULLTEXT01.pdf
2015
Karin Grannas “Improvements and Applications of in situ Proximity Ligation Assays”
http://uu.diva-portal.org/smash/get/diva2:801280/FULLTEXT01.pdf
2016
Linda Arngården “Analysis of signaling pathway activity in single cells using the in situ Proximity Ligation Assay”
http://uu.diva-portal.org/smash/get/diva2:915351/FULLTEXT01.pdf
2018
Axel Klaesson “Development of DNA-based methods for analysis of protein interactions”
http://uu.diva-portal.org/smash/get/diva2:1177746/FULLTEXT01.pdf
2023
Despoina Kermpatsou ”Method development for analysis of protein interactions”
http://uu.diva-portal.org/smash/get/diva2:1712557/FULLTEXT01.pdf
Mattias Leino “Advancing DNA-based proximity methods”
http://uu.diva-portal.org/smash/get/diva2:1740390/FULLTEXT01.pdf
Marie Rubin Sander “Analysis of PDGF receptor internalization and signaling using proximity ligation assays”
http://uu.diva-portal.org/smash/get/diva2:1742559/FULLTEXT01.pdf
Publikationer
Ingår i Journal of Steroid Biochemistry and Molecular Biology, 2024
- DOI för Cellular responses to silencing of PDIA3 (protein disulphide-isomerase A3): Effects on proliferation, migration, and genes in control of active vitamin D
- Ladda ner fulltext (pdf) av Cellular responses to silencing of PDIA3 (protein disulphide-isomerase A3): Effects on proliferation, migration, and genes in control of active vitamin D
The method developer's guide to oligonucleotide design
Ingår i Expert Review of Proteomics, s. 65-80, 2024
- DOI för The method developer's guide to oligonucleotide design
- Ladda ner fulltext (pdf) av The method developer's guide to oligonucleotide design
Ingår i Nucleic Acids Research, 2024
- DOI för Visualizing DNA single- and double-strand breaks in the Flash comet assay by DNA polymerase-assisted end-labelling
- Ladda ner fulltext (pdf) av Visualizing DNA single- and double-strand breaks in the Flash comet assay by DNA polymerase-assisted end-labelling
Ingår i Biochemical and Biophysical Research Communications - BBRC, s. 195-201, 2023
- DOI för Activated EGFR and PDGFR internalize in separate vesicles and downstream AKT and ERK1/2 signaling are differentially impacted by cholesterol depletion
- Ladda ner fulltext (pdf) av Activated EGFR and PDGFR internalize in separate vesicles and downstream AKT and ERK1/2 signaling are differentially impacted by cholesterol depletion
Large-scale phage-based screening reveals extensive pan-viral mimicry of host short linear motifs
Ingår i Nature Communications, 2023
- DOI för Large-scale phage-based screening reveals extensive pan-viral mimicry of host short linear motifs
- Ladda ner fulltext (pdf) av Large-scale phage-based screening reveals extensive pan-viral mimicry of host short linear motifs
Purification of DNA oligonucleotides to improve hybridization chain reaction performance
Ingår i New Biotechnology, s. 33-40, 2023
- DOI för Purification of DNA oligonucleotides to improve hybridization chain reaction performance
- Ladda ner fulltext (pdf) av Purification of DNA oligonucleotides to improve hybridization chain reaction performance
A method for Boolean analysis of protein interactions at a molecular level
Ingår i Nature Communications, 2022
- DOI för A method for Boolean analysis of protein interactions at a molecular level
- Ladda ner fulltext (pdf) av A method for Boolean analysis of protein interactions at a molecular level
Ingår i Cellular Signalling, 2022
- DOI för Differential impact of lipid raft depletion on platelet-derived growth factor (PDGF)-induced ERK1/2 MAP-kinase, SRC and AKT signaling
- Ladda ner fulltext (pdf) av Differential impact of lipid raft depletion on platelet-derived growth factor (PDGF)-induced ERK1/2 MAP-kinase, SRC and AKT signaling
Ingår i Analytical Chemistry, s. 1842-1850, 2021
Ingår i Mutation research. Genetic toxicology and environmental mutagenesis, 2020
Dynamin inhibitors impair platelet-derived growth factor beta-receptor dimerization and signaling
Ingår i Experimental Cell Research, s. 69-79, 2019
- DOI för Dynamin inhibitors impair platelet-derived growth factor beta-receptor dimerization and signaling
- Ladda ner fulltext (pdf) av Dynamin inhibitors impair platelet-derived growth factor beta-receptor dimerization and signaling
Ingår i Journal of Experimental Biology, 2019
Ingår i Molecular Systems Design & Engineering, s. 1058-1065, 2019
- DOI för Optimization of proximity-dependent initiation of hybridization chain reaction for improved performance
- Ladda ner fulltext (pdf) av Optimization of proximity-dependent initiation of hybridization chain reaction for improved performance
Improved efficiency of in situ protein analysis by proximity ligation using UnFold probes
Ingår i Scientific Reports, 2018
- DOI för Improved efficiency of in situ protein analysis by proximity ligation using UnFold probes
- Ladda ner fulltext (pdf) av Improved efficiency of in situ protein analysis by proximity ligation using UnFold probes
Ingår i Current Protocol in Cytometry, 2017
Flow Cytometric Measurement of Blood Cells with BCR-ABL1 Fusion Protein in Chronic Myeloid Leukemia
Ingår i Scientific Reports, s. 1-9, 2017
- DOI för Flow Cytometric Measurement of Blood Cells with BCR-ABL1 Fusion Protein in Chronic Myeloid Leukemia
- Ladda ner fulltext (pdf) av Flow Cytometric Measurement of Blood Cells with BCR-ABL1 Fusion Protein in Chronic Myeloid Leukemia
Insufficient antibody validation challenges oestrogen receptor beta research
Ingår i Nature Communications, 2017
- DOI för Insufficient antibody validation challenges oestrogen receptor beta research
- Ladda ner fulltext (pdf) av Insufficient antibody validation challenges oestrogen receptor beta research
Ingår i Molecular Diagnostics (Third Edition), s. 219-231, Academia Press, 2017
Antagonists of IGF: Vitronectin Interactions Inhibit IGF-I-Induced Breast Cancer Cell Functions
Ingår i Molecular Cancer Therapeutics, s. 1602-1613, 2016
Ingår i Haematologica, s. 668-669, 2016
Ingår i Scientific Reports, 2016
- DOI för Detecting individual extracellular vesicles using a multicolor in situ proximity ligation assay with flow cytometric readout
- Ladda ner fulltext (pdf) av Detecting individual extracellular vesicles using a multicolor in situ proximity ligation assay with flow cytometric readout
Ingår i Proteomes, 2016
Ingår i Molecular & Cellular Proteomics, s. 1848-1856, 2016
The protein kinase LKB1 negatively regulates bone morphogenetic protein receptor signaling
Ingår i Oncotarget, s. 1120-1143, 2016
- DOI för The protein kinase LKB1 negatively regulates bone morphogenetic protein receptor signaling
- Ladda ner fulltext (pdf) av The protein kinase LKB1 negatively regulates bone morphogenetic protein receptor signaling
Ingår i British journal of virology, s. 11-23, 2016
Closing in on life - proximity dependent methods for life sciences
Ingår i Oncotarget, s. 17867-17868, 2015
Ingår i Scientific Reports, 2015
Crosstalk between Hippo and TGF beta: Subcellular Localization of YAP/TAZ/Smad Complexes
Ingår i Journal of Molecular Biology, s. 3407-3415, 2015
Ingår i Molecular Oncology, s. 503-512, 2015
E-cadherin can limit the transforming properties of activating beta-catenin mutations
Ingår i EMBO Journal, s. 2321-2333, 2015
Functional loss of IκBε leads to NF-κB deregulation in aggressive chronic lymphocytic leukemia
Ingår i Journal of Experimental Medicine, s. 833-843, 2015
- DOI för Functional loss of IκBε leads to NF-κB deregulation in aggressive chronic lymphocytic leukemia
- Ladda ner fulltext (pdf) av Functional loss of IκBε leads to NF-κB deregulation in aggressive chronic lymphocytic leukemia
Next-Generation Pathology: Surveillance of Tumor Microecology
Ingår i Journal of Molecular Biology, s. 2013-2022, 2015
Proximity-dependent initiation of hybridization chain reaction
Ingår i Nature Communications, 2015
- DOI för Proximity-dependent initiation of hybridization chain reaction
- Ladda ner fulltext (pdf) av Proximity-dependent initiation of hybridization chain reaction
Analysis of protein interactions in situ by proximity ligation assays
Ingår i High-Dimensional Single Cell Analysis, s. 111-26, Springer, 2014
Ingår i PLOS ONE, 2014
- DOI för Fine-Tuning of Smad Protein Function by Poly(ADP-Ribose) Polymerases and Poly(ADP-Ribose) Glycohydrolase during Transforming Growth Factor β Signaling
- Ladda ner fulltext (pdf) av Fine-Tuning of Smad Protein Function by Poly(ADP-Ribose) Polymerases and Poly(ADP-Ribose) Glycohydrolase during Transforming Growth Factor β Signaling
Ingår i Blood, 2014
Ingår i PLOS ONE, 2013
- DOI för Association of the Protein-Tyrosine Phosphatase DEP-1 with Its Substrate FLT3 Visualized by In Situ Proximity Ligation Assay
- Ladda ner fulltext (pdf) av Association of the Protein-Tyrosine Phosphatase DEP-1 with Its Substrate FLT3 Visualized by In Situ Proximity Ligation Assay
Ingår i Nature Protocols, s. 355-372, 2013
Parallel Visualization of Multiple Protein Complexes in Individual Cells in Tumor Tissue
Ingår i Molecular & Cellular Proteomics, s. 1563-1571, 2013
Ingår i Journal of Pathology, s. 705-718, 2013
Ingår i New Biotechnology, s. 144-152, 2013
Ingår i Oncogene, s. 3606-3615, 2013
Ingår i European Journal of Human Genetics, s. 301-309, 2013
Ingår i Arteriosclerosis, Thrombosis and Vascular Biology, s. 1255-1263, 2012
Ingår i Journal of Cellular and Molecular Medicine (Print), s. 1474-1484, 2012
Ingår i Molecular & Cellular Proteomics, 2012
Ingår i The EPMA journal, 2012
Molecular tools for companion diagnostics
Ingår i New Biotechnology, s. 634-640, 2012
Ingår i Journal of Pathology, s. 378-390, 2012
Visualising individual sequence-specific protein-DNA interactions in situ
Ingår i New Biotechnology, s. 589-598, 2012

