Linus Sandegrens forskargrupp

Dynamik hos plasmidburen antibiotikaresistens
Om du vill jobba hos oss, kontakta Linus Sandegren
Vi studerar hur bakterier utvecklar antibiotikaresistens och bakteriell virulens för att bättre förstå vilka faktorer och molekylära mekanismer som påverkar evolutionen av dessa. Vi är en del i en mycket dynamisk forskningsmiljö med ett flertal forskargrupper som jobbar med komplementära frågeställningar och metoder och vi samarbetar med forskare på flera andra universitet och sjukhus.
Dynamik hos plasmidburen antibiotikaresistens
Vi studerar fundamentala aspekter av hur resistensplasmider etableras och sprids mellan patogena bakterier och hur de fungerar sop plattformar för evolution av antibiotikaresistens. Huvudfokus ligger på att förstå hur faktorer som stabilitet, mobilitet, positive selektion och eventuella kostnader för cellen påverkar hur framgångsrika resistensplasmider blir i bakteriella populationer. Vi använder oss främst av experimentella system baserade på kliniska multiresistensplasmider som kodar för extended-spectrum β-lactamases (ESBLs) i Gramnegativa bakterier (Escherichia coli och Klebsiella pneumoniae). Dessa plasmider utgör ett ökande kliniskt problem genom att de förser bakterierna med resistensgener mot den viktigaste gruppen av antibiotika - β-laktamer.
Vi har fyra huvudfrågeställningar inom de här projekten:
- Hur påverkar låga antibiotikakoncentrationer spridning, selektion och stabiliteten av multiresistensplasmider?
- Vilka plasmidfaktorer ger en kostnad för bakteriecellen och hur kan bakterien kompensera för kostnaden att bära på plasmider?
- Hur vanliga är genamplifieringar av resistensgener på plasmider och hur påverkar dynamiken hos amplifieringar evolutionen av ny resistens?
- Hur påverkar bakteriell biofilmsbildning utvecklingen av resistens och spridning av resistensplasmider?
Genom dessa studier lär vi oss mer om hur bakteriella celler och plasmider evolverar tillsammans och hur selektion av ny resistens kan accelereras genom kopietalsförändringar av resistensgener på plasmider. Genom bättre förståelse av de evolutionära mekanismerna och molekylära förändringar som resistenta bakterier genomgår så kan vi designa våra antibiotikabehandlingar så att de minskar uppkomsten av resistens.
Kombinatoriska effekter av plasmidburna β-laktamaser leder till resistens mot behandling med β-laktamasinhibitorer och β-laktamantibiotika
Den främsta resistensmekanismen mot β-laktamer hos Gramnegativa bakterier är produktion av β-laktamaser, enzymer som bryter ned antibiotikan. Genom att kombinera β-laktamantibiotika med ämnen som blockerar dessa enzymer, så kallade β-laktamasinhibitorer, kan behandlingseffekten fås tillbaka. Tyvärr kan bakterierna utveckla resistens genom att överuttrycka sina β-laktamaser eller genom att minska insläppet av antibiotika och inhibitor i cellen genom att minska uttryckt av poriner i yttermembranet. Det är vanligt att bakterier bär på gener för flera olika β-laktamaser på samma plasmid och vi studerar hur denna kombination av β-laktamaser påverkar utvecklingen av resistens mot β-laktam/β-laktamasinhibitor kombinationer. Vi fokuserar särskilt på hur skillnader i katalytiskt spektrum hos de olika β-laktamaserna påverkar bakteriens möjlighet att snabbt utveckla resistens mot olika kombinationer av behandling genom amplifiering av de respektive generna på plasmiderna.
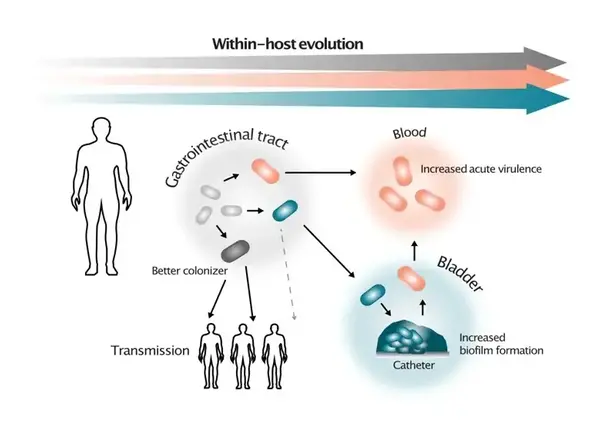
Evolutionära scenarier för urval av subpopulationer inom värd med förändrade virulens. Illustrationer av Greta Zaborskyte.
Evolution av Klebsiella pneumoniae inom patienter - Hur specialiserar sig en opportunistisk patogen?
Hur bakterier balanserar mellan att kolonisera oss utan att ge några symtom eller att orsaka infektioner är en central fråga inom infektionsbiologin. Trots mycket forskning runt specifika virulensfaktorers roll i infektionsförloppet saknar vi fortfarande mycket kunskap när det gäller de evolutionära selektionstryck och spontana genetiska förändringar som leder till kortsiktiga förändringar i bakteriers interaktion med värden. En nyckelfråga är hur balansen mellan optimal fitness under kolonisation och förmågan att infektera oss påverkar bakteriers förmåga att associeras med en värdorganism på kort och lång sikt. I det här projektet kombinerar genotypiska och fenotypiska studier av en unik kollektion av Klebsiella pneumoniae isolat från ett stort sjukhusutbrott av multiresistenta bakterier och samlingar av globala högrisk-kloner för att utröna vad som särskiljer bakterier som orsakar utbrott. Genom experimentell evolution, avancerad bakteriegenetik och nya biofilmsmodeller i laboratoriemiljö kan vi beskriva och testa kritiska steg i evolutionen mellan värd och patogen. K. pneumoniae är vanligen en opportunistisk patogen, men på senare tid har hypervirulenta kloner börjat dyka upp i globalt. Det har föreslagits att evolutionen av dessa har skett genom parallell utveckling av resistens och virulens.
Biofilmsevolution och plasmiddynamik hos Klebsiella pneumoniae
Bakterier kan växa som samlade populationer på ytor och kallas då för biofilmer. Biofilmsväxt är ett stort problem i samband med kroniska infektioner och infektioner på inplantat som höftleder, hjärtklaffar eller katetrar. Ett stort problem med biofilmer är att bakterierna i biofilmen är mer skyddade mot både immunförsvaret och mot behandling med antibiotika. Bakterier tar sig ofta in i kroppen genom olika typer av infarter och katetrar. För opportunistiska K. pneumoniae brukar det resultera i urinvägsinfektioner eller lunginflammationer hos sjukhusvårdade patienter. I biofilmen ligger bakterierna dessutom tätt tillsammans vilket i vissa fall har visats leda till ökad spridning av plasmider mellan cellerna, medans det i andra fall har visats leda till mindre spridning. Biofilmen är också en miljö där bakterierna är uppdelade i olika mindre kluster vilket kan påverka selektionen av resistens genom att olika bakterier utsätts för olika mycket antibiotika beroende på var de befinner sig och vilka andra bakterier som finns runt om.
Här studerar vi hur K. pneumoniae kan utveckla sin biofilmsbildande förmåga och vilka molekylära mekanismer som ligger bakom det. Genom experimentell evolution i ytbundna biofilmer kan vi imitera hur bakterier bildar biofilmer på tex katetrar. Vi använder också dessa system för att studera hur strukturen i biofilmer påverkar hur resistensplasmider sprids och hur resistensevolutionen påverkas. Vi vill på detta sätt förstå hur antibiotikabehandlingar, speciellt vid låga koncentrationer, påverkar bakterierna i biofilmen och hur interaktionen mellan bakterier och plasmider bidrar till resistensutveckling och spridning av resistensgener.
Vi har utvecklat ett speciellt system för att studera evolution av biofilmer i laboratoriet som också är bra för att kunna testa hur bakterier i biofilm påverkas av olika typer av antibiotikabehandlingar. Systemet är kompatibelt med in situ mikroskopi där man kan följa cellerna under förloppet. Därigenom kan vi följa fluorescent märkta bakterier och se hur plasmider kan spridas inom biofilmen genom speciella genetiska system. Kunskapen som genereras i de här projekten kan användas för att förbättra behandlingen av biofilms-associerade infektioner.
CAD filer för FlexiPeg-biofilmsystemet kan laddas ned här.
